| Zecken HLFS Ursprung 2006 |
|
| Startseite | |
| Zusammenfassung | |
| Motivation | |
| Zecken | |
| Jagd | |
| Laboranalytik | |
| Auftrag | |
| Video | |
| Videoblog | |
| Literaturverzeichnis | |
| Pädagogik | |
| Infoblatt | |
| Team | |
| Impressum | |

![]()
 |
|
| Damit die Enzyme einwirken können, brauchen sie Zeit und die richtige Temperatur. |
 |
|
| F.I.T. - Voller Vorfreude. |
 |
|
| Konzentration auf höchster Stufe. |
 |
|
| Verdammt schwierig, aber schaffbar. |
 |
|
Bei über 20 Reagenzien ist Ordnung das Um und Auf. |
 |
|
| Sorgfältige Desinfektion vor und nach jedem Arbeitsschritt. |
 |
|
| Verbrauchtes Material wurde autoklaviert. |
 |
|
| Arbeiten mit Mikro-Pipetten, eine große Herausforderung. |
Laboranalytik
Nachweis von FSME-Viren und Borrelien in Zecken
Die Erreger der Borreliose sind Bakterien, so genannte Spirochäten (Schraubenbakterien). Das Erbgut der Borrelien ist DNA.
Der Erreger der FSME ist ein Virus. Das Erbgut der FSME-Viren ist RNA.
Inzwischen ist es möglich geworden, sowohl den FSME- als auch den Borreliose-Erreger in der Zecke direkt durch einen DNA-Test nachzuweisen.
Dieser Test liefert im Gegensatz zu serologischen Laboruntersuchungen, z.B. dem ELISA-TEST zum Nachweis von Antikörpern nach einigen Wochen im Blut des Opfers, ein sehr schnelles Ergebnis und im Ernstfall kann sofort mit einer speziellen Therapie begonnen werden.
Für das Schullabor werden die gefangenen Zecken mehrere Wochen in 70%igem Alkohol bei -70°C gelagert. Der Alkohol tötet in dieser Zeit verlässlich alle Krankheitskeime ab, sodass uns bei der Laborarbeit keinerlei Gefahr droht und die Lagerung bei diesen extrem niedrigen Temperaturen garantiert, dass das Erbgut der Erreger nicht durch Enzyme abgebaut wird. Ein professionelles Labor würde die Zecken sofort verarbeiten, weil die Mitarbeiter im sicheren Umgang mit gefährlichen Krankheitserregern geübt sind.
Die Identifizierung von Borrelien bzw. der FSME-Viren in der Zecke erfolgt mit Hilfe der DNA/RNA-Analytik, d.h. es wird geprüft, ob die Erbsubstanz des jeweiligen Erregers in der Zecke, also in der Probe, nachweisbar ist.
Das bedeutet für den potentiellen Nachweis, dass aus den gefangenen Zecken nicht nur die Borrelien-DNA, sondern auch die FSME-RNA isoliert werden muss. Normalerweise kann man so genannte Kits kaufen, die bereits ein detailliertes Protokoll zur Isolierung von entweder DNA oder RNA enthalten. Nachdem in diesem besonderen Fall jedoch jede Zecke sowohl auf Borrelien als auch auf FSME-Viren getestet werden sollte, musste ein Isolierungsprotokoll für die gesamten Nukleinsäuren (DNA+RNA) gefunden werden. Nach längerer Suche und mehreren Vortests erwies sich der Kit „MasterPure Complete DNA & RNA“ der Firma Epicentre aus den USA als geeignet:
Als erster Schritt zur Gewinnung des Erregererbgutes wird der Chitinpanzer der Zecken entweder durch Zerreiben in flüssigem Stickstoff oder mithilfe eines Ultraturrax, das ist ein spezieller kleiner „Hochleistungspürierstab“ mit über 20.000 Umdrehungen pro Minute, mechanisch zerstört und die Zellen anschließend mit Enzymen (Proteinasen) behandelt und aufgeschlossen (lysiert). Mit mehreren aufeinander folgenden Reinigungsschritten werden störende Fette, Proteine, Zellorganellen und Salze entfernt. Nach der Isolierung der Erbsubstanz erfolgt der Nachweis der potentiell vorhandenen Borrelien-DNA bzw. FSME-Virus-RNA mithilfe der so genannten PCR-Methode.
Die Polymerase-Kettenreaktion (PCR) spielt heutzutage eine wichtige Rolle in der Routinediagnostik zum Nachweis von Bakterien oder Viren und es erlaubt die spezifische Vervielfältigung geringster DNA-Mengen des gesuchten Erregers.
Der Nachweis von Borrelien ist relativ einfach mit einem PCR –Schritt möglich, da deren Erbgut aus DNA besteht. Eine geeignete Sequenz zur Vervielfachung eines Borrelien-Gens wurde aus der wissenschaftlichen Literatur ausgewählt.
Im Falle des FSME-Nachweises muss die Viren-RNA zuerst in DNA umgeschrieben werden (reverse Transkription), um danach in einem „nested PCR-Verfahren“ vervielfältigt zu werden. Bei der „nested-PCR“ werden 2 PCR-Durchgänge hintereinander durchgeführt, damit auch geringste Mengen der gesuchten Information aufgespürt werden können. Bei der 2. PCR-Reaktion wird dabei ein etwas kleineres Fragment des 1. Durchgangs vervielfältigt. Die „nested PCR“ erhöht also die Sensitivität und Spezifität des Nachweises.
Dieses Verfahren wird als RT-PCR ( Reverse Transkriptase - Polymerase Kettenreaktion) bezeichnet: Zuerst wird eine Reverse Transkriptase (RT) eingesetzt, die die RNA in cDNA (c steht für komplementär) umschreibt. Diese cDNA kann im Anschluss als Ausgangsprodukt in einer PCR verwendet werden, um die gesuchten Sequenzen zu vervielfältigen.
Die Produkte der PCR werden dann auf einem Agarosegel analysiert, wodurch die DNA–Fragmente nach der Größe sortiert und ausgewertet werden können.
Die nachfolgende Interpretation der Resultate ist vergleichsweise einfach. Ein negatives PCR-Ergebnis besagt, dass kein Erregererbgut in der Zecke gefunden werden konnte und somit das Risiko einer Infektion nahezu ausgeschlossen ist. Erhält man jedoch ein positives PCR-Ergebnis, so beweist dies das Vorhandensein von Borrelien bzw. FSME-Viren in der Zecke und damit das Risiko einer erfolgten Übertragung der Krankheitskeime im Verlaufe des Zeckenstichs.
Um sicher zu gehen, dass im Lauf der diagnostischen Untersuchung kein Fehler passiert ist, wurde zusätzlich eine Positivkontrolle durchgeführt. RNA aus den Mitochondrien der Zecken wurde bei jeder Probe ebenfalls in cDNA umgeschrieben (reverse Transkription) und im Anschluss daran mit einer PCR vervielfältigt. Um eine verlässliche Aussage treffen zu können, mussten demnach alle Kontrollreaktionen ein sichtbares PCR-Produkt liefern, wohingegen im Falle der Borrelien- und FSME-PCR nur infizierte Zecken ein Signal am Agarosegel liefern sollten. Im Falle eines Fehlers im Laborablauf würde auch keine Positivkontrolle nachweisbar sein und zeigen, dass der Versuch wiederholt werden müsste.
Das ganze Nachweisverfahren wurde mit originaler Borrelien-DNA, FSME–RNA und FSME c-DNA sorgfältig überprüft. Dr. Stephan Aberle, Klinisches Institut für Virologie der Medizinische Universität Wien, stellte uns die Referenzproben vom FSME-Erbgut zur Verfügung, Dr. Arnold Bito, Fachbereich Zellbiologie der Universität Salzburg die Referenzproben zum Borrelienerbgut.
1. Der Testablauf im Überblick:
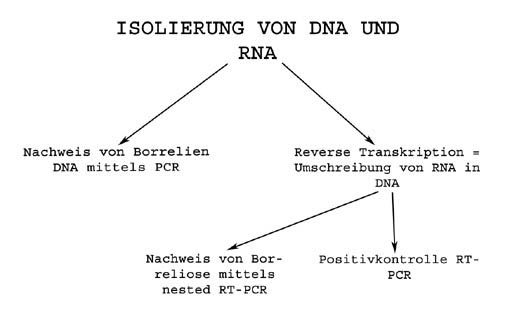
· Isolierung von DNA und RNA aus den Zecken
· Selektiver Nachweis von Borrelien DNA mittels Polymerase-Kettenreaktion (PCR)
· Reverse Transkription (Umschreibung) der isolierten RNA in cDNA (komplementäre DNA)
· Selektiver Nachweis von FSME-viraler cDNA mittels Polymerase-Kettenreaktion (PCR)
· Nachweis von Zecken-cDNA als Positivkontrolle
· DNA-Gelelektrophorese zur Sichtbarmachung der PCR-Produkte und Auswertung der Ergebnisse
2. Die experimentelle Vorgangsweise – Das Laborprotokoll
2.1 DNA und RNA Isolierung
(MasterPure Complete DNA and RNA Purification Kit, Fa. Epicentre)
· Einige Zecken in flüssigem Stickstoff aufbrechen oder im Ultraturrax zerkleinern
· Zugabe von 300µl Tissue and Cell Lysis Solution
· Zugabe von 1µl Proteinase K
· vortexen
· 15min bei 65°C inkubieren, dabei alle 5min vortexen (Zellen werden aufgebrochen)
· 3min auf Eis stellen
· Zugabe von 160µl MPC Protein Precipitation Solution (Proteine werden zerstört und fallen aus)
· 10sec vortexen
· 10min zentrifugieren (damit werden wir die Proteine los)
· Überstand (enthält die Nukleinsäuren) in ein neues Röhrchen überführen
· Zugabe von 500µl eiskaltem Isopropanol, 30-40x Röhrchen kippen zum Mischen
· 10min zentrifugieren (Nukleinsäuren sind jetzt im Pellet!)
· VORSICHTIG den Alkohol ableeren
· Pellet (DNA und RNA!!!) mit 150µl 75% EtOH waschen
· Alkohol vollständig abheben, pellet kurz trocknen lassen
· DNA und RNA in 35µl Wasser lösen.
2.2 Vervielfältigung der Borrelien DNA mittels PCR
(Primer:Priem S, Rittig MG, Kamradt T, Burmester GR, Krause A.
An optimized PCR leads to rapid and highly sensitive detection of Borrelia burgdorferi in patients with Lyme borreliosis.
J Clin Microbiol. 1997, 35(3):685-90.
Protokoll zur Vervielfältigung des Borrelien Gens p66:
Produkt hat @ 400 bp
ddH2O 36.8 µl
PCR buffer 10x 5 µl
dNTPs 1 µl 10 mM
Primer 1 1 µl 10 pmol/µl
cgaagatactaaatctgt
Primer 2 1 µl 10 pmol/µl
gatcaaatatttcagctt
Probe (DNA) 1 µl MgCl2 4 µl 25 mMTaq polymerase 0.2 µl 2U/µl
PCR-Bedingungen:
initial 3 min 94°C,
Dann 35 Cyclen:
94 °C.......30 sec
45 °C.......30 sec
72 °C.......30 sec
2.3 Vervielfältigung der FSME RNA und der Positivkontrolle mittels RT – PCR
(RevertAid First Strand cDNA Synthesis Kit, Fa. Fermentas)
Im ersten Schritt wird die RNA durch Reverse Transkription in cDNA umgeschrieben. Eine Unmenge an kurzen zufallsgenerierten Primern garantiert, dass sämtliche vorhandene RNA, sowohl FSME Virenspezifische als auch Zecken-interne für die Positivkontrolle transkribiert wird.
5 µl Probe (RNA)
1 µl random hexamer primer
6 µl DEPC-behandeltes Wasser (enthält sicher keine RNA-sen!)
· Kurz vortexen und 3sec abzentrifugieren
· 5min 70°C, kurz auf Eis, abzentrifugieren
4 µl reaction buffer
1 µl Ribonuclease Inhibitor
2 µl dNTPs
· Kurz vortexen und 3sec abzentrifugieren
· 5min Raumtemperatur
· Zugabe von 1 µl Reverse Transcriptase
· 10min Raumtemperatur
· 60min 42°C
· Stop der Reaktion durch 10min 70°C
Im zweiten Schritt schließt sich eine normale PCR an, in der die eben erzeugte cDNA als Template (Vorlage) verwendet wird. Erst bei diesem Schritt wird selektiv mit passenden Primern die gesuchte cDNA Sequenz amplifiziert
Es wird
a.) FSME-Virus RNA (res. cDNA) und
b.) als Positivkontrolle mitochondriale RNA (res. cDNA) von Zecken
vermehrt
a) Die Vervielfältigung der FSME cDNA
(Primer von Dr. Stephan Aberle, Klinisches Institut für Virologie, Medizinische Universität Wien)
Um die Spezifität und Sensitivität des FSME- Nachweises zu erhöhen, wird eine sog. nested-PCR angesetzt. Hierbei wird in einem ersten Schritt die PCR mit dem Template über einige Zyklen durchgeführt. Anschließend werden in einer zweiten Runde neue Primer eingesetzt. Die neuen Primer liegen weiter innen als die ersten, sodass in diesem zweiten Schritt nur noch die spezifischen DNA Abschnitte des ersten Schrittes amplifiziert werden. Dies erhöht die Empfindlichkeit und die Effizienz so enorm, dass sehr sorgfältig und kontaminationsfrei gearbeitet werden muss, da Verunreinigungen ungewünschte Auswirkungen haben können.
Bei der nested PCR wird also nach erfolgreicher erster Amplifikation das erste PCR Produkt als Template für eine zweite PCR verwendet.
1. PCR FSME:
10 µl cDNA (aus 2,3)
1 µl primer1
(5´-gaggctgaacaactgcacga-3´)
1 µl primer2
(5´-gaacacgtccattcctgatct-3´)
1.5 µl dNTPs (10mM)
0.2 µl Taq Polymerase
5 µl Puffer
4 µl MgCl2
27.3 µl H2O
25 Zyklen:
94 °C.......30 sec
40 °C.......30 sec liefert ein Produkt von 357 bp
68 °C.......30 sec
2. PCR FSME:
2 µl der ersten PCR
1 µl primer3
(5´-acggaacgtgacaaggctag-3´)
1 µl primer4
(5´-gcttgttaccatctttggag-3´)
1.5 µl dNTPs
0.2 µl Taq Polymerase
5 µl Puffer
4 µl MgCl2
35.3 µl H2O
30 Zyklen:
94 °C.......30 sec
50 °C.......30 sec liefert ein Produkt von 252 bp
72 °C.......30 sec
b) Die Vervielfältigung der mitochondrialen Zecken cDNA als Positivkontrolle.
(Primer lt. Info von Dr. Judith Kießling, Institut für Medizinische Mikrobiologie, Infektions- und Seuchenmedizin, Tierärztliche Fakultät, Ludwig-Maximilians-Universität München)
Als Kontrolle, ob erstens die Isolierung von RNA und zweitens auch die reverse Transkription funktioniert haben, wird ein bestimmter Abschnitt von mitochondrialer Zecken-RNA, also respektive cDNA, vervielfältigt.:
PCR-Ansatz:
2 µl cDNA (aus 2.3)
1 µl dNTPs
1 µl primer 1
F-16S: aaaaaaatactctagggataacagcgtaa
1 µl primer 2
R-16S: accaaaaaagaatcctaatccaaca
5 µl Puffer
4 µl MgCl2
0.2 µl Taq Polymerase
35,8 µl H2O
35 Zyklen:
94 °C.......30 sec
56 °C.......30 sec liefert ein Produkt von etwa 700 bp
72 °C.......30 sec
2.4 Nachweis aller PCR-Produkte im Agarosegel
Um die PCR-Produkte erstens sichtbar zu machen und zweitens deren Größe zu bestimmen, wird eine Agarose-Gel-Elektrophorese durchgeführt.
DNA-Gel:
1 % Agarosegel in 0,5x Tris-Borat-Puffer pH 8,6 (TBE)
+ 8 µl Farbstoff (Gelstar)
DNA-Farbstoff (Gelstar): Färbereagens, das sich in die DNA einschiebt.
Wird von Ultraviolettlicht (UV) zum Leuchten (= Fluoreszenz) angeregt - und macht so die einzelnen DNA - Fragmente am Gel sichtbar.
Probenvorbereitung für die Elektrophorese:
50 µl Probe (PCR)
10 µl Probenpuffer (BPB)
Probenpuffer: enthält einen Farbstoff (= Bromphenolblau), der es ermöglicht, die Lauffront am Gel zu verfolgen und Glyzerin zur „Beschwerung“ der DNA, die dadurch in die Geltaschen sinkt.
25 µl von den Proben auf den Boden der Geltaschen pipettieren;
Dauer der Trennung mindestens 45 Minuten bei 100 mA
Die DNA wandert vom Minus - zum Pluspol, das entspricht den Kabelfarben Schwarz zu Rot. Anhand der Farbfront kann man die Wanderung der DNA im Gel beobachten. Nach etwa 45 Minuten wird das Gel aus der Kammer genommen und auf den Illuminator gelegt. Auf einem BlaulichtIlluminator wird das Gel betrachtet und fotografiert. Die gefärbten DNA-Banden leuchten grün-gelb.
Der Marker gibt Informationen über die Größe der Banden.
Bei dieser Technik bleiben längere DNA-Stückchen, die durch ein elektrisches Feld wandern, länger im Agarosenetzwerk hängen als kürzere Fragmente und werden somit nach Größe sortiert.